Table of Contents
RNA-seq-based bacterial transcriptome analysis
RNA-seq을 이용한 진핵 생물의 전사체(transcriptome) 분석에서는 TopHat-Cufflioks-Cuffmerge-Cuffdiff-(CummeRbund)로 이어지는 Tuxedo protocol이 표준으로 여겨지고 있다. 여기에서는 splicing을 고려한 read alignment, 새로운 유전자의 발견, novel splice variant의 발견 등이 관건이 된다. Cufflinks version 2.2.0부터는 Cuffquant/Cuffnorm이라는 새로운 프로그램을 포함하는 워크플로우로 바뀌었다(링크).
그러나 이 방법을 박테리아 대상의 RNA-seq data analysis에 그대로 적용하는 것은 무리가 있다. 특히 read alignment 과정이 박테리아에게는 딱 맞지 않는다. 왜냐하면 유전자 밀도가 낮은 진핵 생물과 달리 원핵 생물은 유전자 배열이 매우 촘촘하여 심지어는 서로 겹치기도 하고, splicing이 일어나지 않기 때문이다. 따라서 일반적인 (genome 유래) read mapping program을 그대로 사용하여 reference genome sequence에 붙이는 것이 더 나을 수 있다. 뿐만 아니라 세균의 경우 워낙 그 종류가 많아서 일부 모델 세균을 제외하면 표준적인 reference(annotation 정보 포함)가 없는 경우가 대다수이다. 즉, 분석을 하려는 당사자가 reference 정보를 같이 준비해야 한다는 뜻이다. 이러한 전체적인 과정은 2016년 발표된 한 논문(“SPARTA: Simple Program Automated reference-based bacterial RNA-seq Transcriptome Analysis)의 그림에 아주 잘 표현되어 있다.
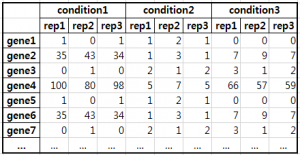
아주 쉽게 이야기하자면 RNA-seq read의 mapping을 통해서 샘플로부터 다음 그림과 같은 형태의 데이터를 얻어내는 것이 첫번째 단계라고 할 수 있다. 각 셀을 채우는 수치는 특정 샘플(조건 및 반복)의 유전자가 갖는 expression value이다. 이것은 read count일 수도 있고, RPKM/FPKM 및 이에 상응하는 값일 수도 있다.
이러한 데이터가 얻어진다면 R 환경에서 돌아가는 다양한 도구 혹은 CLC Genomics Workbench 등을 사용하여 조건의 조합에 대한 DEG set를 산출할 수 있게 된다.
Gene annotation file의 호환성
GenBank flat file
CLC Genomics Workbench를 제외하면 GenBank flat file은 보통 RNA-seq analysis program에서 reference sequence + annotation의 정보 제공 용도로 잘 쓰이지는 않는다. 그러나 임포트한 상태 그대로가 아니라 Track Tools → Convert to Tracks를 사용하여 sequence track 및 annotation track으로 전환해야 된다. 여기서 문제가 되는 것은 원핵 미생물 유래 표준 GenBank file(available from NCBI)의 protein-coding gene에 대해서는 오직 gene과 하위 CDS feature만이 존재한다.
RAST server와 Prokka에서 만든 GenBank file에는 gene feature가 없이 CDS/rRNA/tRNA 등의 subfeature만 존재한다. 따라서 적당한 스크립트를 작성하여 상위 feature인 gene을 생성해야 한다.
어떤 경우든 mRNA feature가 없으면 CLC Genomics Workbench에서 mRNA track을 뽑아내지 못하게 되고, 결과적으로 RPKM 값은 계산이 되지 않는다(TPM, total gene reads 및 unique gene reads 값은 만들어진다). 따아서 GFF 파일을 조작하여 사용하는 것이 훨씬 낫다(아래 참조)
추가 정보 CLC Genomics Workbench의 RNA-seq Analysis 옵션에 “Calculate RPKM for genes without transcript”가 있음을 발견하였다. 그렇다면 mRNA feature 없이 gene만 존재하여도 RPKM 계산이 가능함을 의미한다. — Haeyoung Jeong 2017/02/23 11:42 물론 이것은 prokaryote의 경우에 한한다. — Haeyoung Jeong 2017/04/04 16:33
추가 정보 GFF3 file을 CLC에서 작업하는 경우 transcript feature를 mRNA로 바꾸어야 나중에 제대로 track으로 표현된다. — Haeyoung Jeong 2017/04/07 18:23
.ptt & .rnt files
과거에는 NCBI의 RefSeq 자료에 ptt/rnt 파일이 존재하였으나 이제는 더 이상 제공되지 않는다. 박테리아의 RNA-seq 데이터 분석용 프로그램인 EDGE-pro와 Rockhopper2 는 아직도 이것을 필요로 하므로, GenBank 파일을 사용하여 적당히 ptt/rnt 파일을 만들어야 한다.
GFF/GTF/GFF3 file
GFF file은 워낙 형식이 느슨하므로 RNA-seq data analysis용 프로그램이 이를 잘 받아들이는지를 사전에 점검해야 한다. 예를 들어 Prokka annotation tool이 만들어낸 GFF3 파일은 뒷부분에 FASTA sequence가 붙어있는데, SPARTA(htseq-count 단계)에서는 에러를 유발한다. 그러나 Ensembl에서 제공하는 GTF/GFF3 file은 특별한 조작을 가할 필요가 없다.
CLC Genomics Workbench에서 이 파일을 사용하려면(non-Ensemble)
- Annotate with GFF file 플러그인을 설치한다.
- Sequence fasta file을 standard import로 불러들인다.
- GFF 파일을 조작하여 gene/mRNA feature를 부가한다($ modifyGFFforCLC_GW.pl original_gff > modified_gff). repeat_region에 상위 gene feature를 추가하지 않도록 주의한다.
- Toolbox에서 Classical Sequence Analysis → General Sequence Analysis → Annotate with GFF/GTF/GVF file을 실행한다. 이때 2. Set parameters에서 “Name handling”을 적절히 수정한다. Name이란 각 feature에 부가되는 유일한 식별자로서 특별히 지정하지 않으면 Name - Gene_name - Gene_ID - Locus_tag - ID의 우선 순위로 적용된다. 작업할 GFF 파일을 열어보고 결정하기 바란다.
- Toolbox에서 Track Tools → Convert to Tracks를 실행한다.
- 이후의 RNA-seq Analysis에서 필요한 annotation track을 고른다. 'Eukaryote' setting에서는 reference sequence와 gene track 및 mRNA track이 전부 필요하고, 'prokaryote' setting에서는 reference sequence와 gene track만 고를 수 있다. Default prokaryote setting에서는 RPKM이 계산되지 않지만, mRNA track이 존재한다면 계산 가능하다.
직접 read count를 추출하려면(htseq-count)
Bowtie2 등으로 만든 SAM 파일이 있다면 HTSeq 유틸리티의 htseq-count를 다음과 같이 실행하여 직접 read count를 추출할 수 있다. htseq-count는 Ensemble GTF 파일을 기준으로 만들어진 것이라서 exon feature를 필수로 요구하며, gene_id를 feature ID로 인식한다. 다음의 실행 사례에서는 CDS와 ID를 사용하도록 옵션을 준 것이다. GFF(3) 파일도 사용 가능하나 호환성을 점검해 볼 필요가 있다.
$ htseq-count -t CDS -i ID alignment.sam annotation.gtf > read_count_per_gene.txt
Count file의 병합
SAM file이 여러개라면 이에 상응하여 같은 수의 read count file이 만들어진다. 이를 하나로 병합하려면 어떻게 하면 좋을까? 엑셀을 사용해도 좋지만, 다음과 같은 간단한 R 코드를 권장한다.
> S1 = read.table('countFile1', row.names=1)
> colnames(S1) = 'S1'
> S2 = read.table('countFile2', row.names=1)
> colnames(S2) = 'S2'
... # 전체 파일 수만큼 반복
> alldata = cbind(S1, S2, S3...)
> write.csv(alldata, file='mergedCountFile.csv')
또는
> write.table(alldata, file='merged.txt', sep="\t")
alldata 데이터프레임에 모든 자료가 채워졌으므로 여러 가지의 조작을 할 수 있다. 이 페이지가 R 기초를 공부하기 위해 만들어진 것은 아니나, 복습을 하는 의미에서 몇 가지를 예시해 보자.
> summary(alldata) > alldata[apply(alldata, 1, sum) < 5,] # 모든 sample의 count를 더해도 5가 안되는 유전자의 출력 > nrow(alldata[apply(alldata, 1, sum) < 5,]) # 위와 같은 row(유전자)가 총 몇개인가? > F.alldata = alldata[apply(alldata, 1, sum) >= 5, ] # 위와 같은 유전자를 제외한 나머지만 추출
다음으로는 bacteria용 RNA-seq data analysis program의 활용 시 유의해야 할 점을 기록해 보았다. 다음의 프로그램은 multiple chromosome/plasmid에 대해서도 잘 동작한다고 매뉴얼에는 나오지만, 여러 개의 contig로 구성된 genome에 대해서는 오류를 발생하는 경우가 많았다. 따라서 sequence gap(NNNN..N)을 사이에 넣어서 적절히 concatenation한 pseudomolecule을 만들어서 genome sequence file 및 gene annotation file을 준비하기를 권장한다.
EDGE-pro
- EDGE-proL Estimated Degree of Gene Expression in Prokaryotic Genomes. Evol. Bioinform. 2013l 9:127-36
- Program website: http://ccb.jhu.edu/software/EDGE-pro/
Bacterial genome의 특성에 맞도록 각 유전자 expression level을 추정하는 (거의) 최초의 프로그램이다. Gene overlap을 고려하여 고유한 방법으로 산출한 read count를 사용, RFKM 값을 계산하는 것이 특징이다. 단, 여러 샘플 간의 differential expression을 계산하지는 않는다. 대신 DESeq에 사용할 수 있도록 출력물을 전환하는 스크립트(edgeToDeseq.perl)을 포함한다. Low-level background를 줄이기 위하여 RPKM < 3이면 0으로 출력한다(사용자 변경 가능). 실행 사례는 다음과 같다. 복수의 choromosome/plasmid가 있는 경우 genome.fa, gene.ptt 및 rna.rnt 내에서 서열의 순서가 동일해야 함에 유의하라.
$ cd example # 설치 디렉토리 하위의 example로 이동 $ ../edge.pl -g genome.fa -p gene.ptt -r rna.rnt -u read_1.fastq -v read_2.fastq -s .. -o out
Replicate가 존재하면 각각에 대하여 따로 실행하면 된다. 가장 중요한 결과 파일은 out.rfkm_0이다. 서열이 여러개이면 _1, _2,.. 순으로 증가한다.
Rockhopper2
- Rockhopper: Computational analysis of bacterial RNA-Seq data. Nucl. Acids. Res. 2013l 41(14):e140
- Rockhopper 2: De novo assembly of bacterial transcriptomes from RNA-seq data. Genome Biol. 2015; 16:1
- Program website: http://cs.wellesley.edu/~btjaden/Rockhopper/
Rockhopper2는 de novo transcript assembly와 operon 예측이 가능하다는 점이 특징인 GUI 프로그램이다. 결과물은 IGV와 연동되어 쉽게 시각화된다. 대부분의 프로그램이 RNA-seq read mapping 단계에서 bowtie2를 사용하는 것이 반하여 자체적으로 개발된 aligner가 내장되어 있다. Expression value와 differential gene expression test를 거친 결과물은 *_transcripts.txt에, operon 구조는 *_operons.txt 파일에 각각 수록된다. 제공되는 expression level은 read number로 나누지 않고 upper quartile로 나누어서 얻은 것이다. Rockhopper2의 소박한 GUI를 소개한다.
*_transcripts.txt의 컬럼
- Transcription Start, Translation Start, Translation Stop, Transcription Stop, Strand
- Name (locus_tag), Synonym, Product
- [Raw Counts, Normalized Counts, RPKM,] Expression ⇐ 조건마다 반복
- pValue, qValue
Replicate가 존재하면 [Raw Counts, Normalized Counts, RPKM] 컬럼은 표시되지 않는다. 개별 반복 샘플에 대한 raw read count 값은 표시되지 않고 조건에 따라 expression 수치만 제공되므로 만약 다른 DEG analysis tool을 사용하려면 각 반복 샘플을 별도로 분석하면 된다. 단, 최소 2 조건을 맞추지 않으면 Expression만 표시됨을 확인하였다.
SPARTA
- SPARTA: Simple Program for Automated reference-based bacterial RNA-seq Transcriptome Analysis. BMC Bioinformatics 2016; 4:17-66
Read 전처리(trimmomatic) 및 QC(fastQC), mapping(bowtie), expression level 추정(HTSeq) 및 dfferential expression analysis(edgeR)를 일괄적으로 실행하는 workflow 방식의 프로그램이다. 일루미나 기반의 sinlge-end read만을 사용할 수 있으므로, pair-end read를 가진 경우에는 forward read file만을 사용하라. DEG 계산에는 edgeR이 쓰인다. 결과 디렉토리에는 DEG 계산과 diagnostic plot을 그리는데 사용한 R 스크립트가 남게 되므로 추후 활용하기에 좋다. 이상에서 소개한 3개 프로그램 중에서 유일하게 전처리 및 QC 도구를 포함하고 있고, 가장 활용성이 높다고 판단된다.
주의 사항
입력 파일의 위치, 조건 수, 각 조건에 따른 샘플 설정 등을 명령행 인터페이스에서 대화식으로 입력하면 된다. 쉬운 설치와 사용을 표방하고 있지만 우분투가 아닌 CentOS에서는 그렇지만도 않다. install_dependencies.sh 스크립트(아래 참조)를 사용하여 필요한 프로그램들을 사전에 설치해 준다고는 하지만 apt-get을 쓰도록 되어있기 때문이다.
#!/bin/bash #Install Python development headers, NumPy, Java, and R sudo apt-get update sudo apt-get install build-essential python2.7-dev python-numpy default-jre r-base r-base-dev sudo R --vanilla --slave < install_edger.r
설치 위치는 반드시 ~/Desktop/SPARTA_Linux-master이어야 한다. 그리고 fastq file 이름에 밑줄이 들어있으면 edgeR 실행 단계에서 에러가 나므로 미리 적절히 바꾸는 것이 좋다. Replicate가 없으면 read count file까지는 생성되지만 역시 edgeR이 실행되지 않으므로 유의하라. GFF 파일로부터 참조하는 feature의 기본 타입과 ID는 exon과 gene_id이므로, 실제 상황에 맞게 수정하여 사용한다. 다음 예를 참조한다.
$ python SPARTA.py --type=CDS --idattr=locus_tag
CLC Genomics Workbench
'Experiment' 설정을 통해서 분석을 수행하는 classical transcriptomics analysis와 Advanced RNA-Seq 플러그인이 제공하는 분석 중에서 적당한 것을 골라서 사용하면 된다. 외부에서 만들어진 read count data(csv 포맷의 텍스트 파일)을 임포트하여 사용하려면 전자를 택한다. Advanced RNA-Seq 플러그인을 설치하면 Toolbox의 Transcriptomics Analysis → RNA-Seq Analysis에 다음과 같이 빨강색 상자로 표시한 새로운 기능이 추가된다. Advanced RNA-Seq 플러그인에서는 track과 metadata file이 매우 중요하게 다루어짐을 기억해 두자.
What's next?
이상의 방법을 통해서 DEG, 즉 서로 다른 조건에 대한 차등 발현 유전자의 목록을 얻었다고 가정하자. 그 다음으로는 어떤 심층 분석을 하면 좋을까? 이 모든 질문에 대한 대답을 여기에서 다 작성할 수는 없는 노릇이다. 비슷한 고민을 하는 연구자의 순수한 질문과 멋진 대답, 그리고 최신 리뷰를 하나 소개하는 것으로 갈음할까 한다.
- A survey of best practices for RNA-seq data analysis. Genome Biol. 2016; 17:13