Table of Contents
R을 이용하여 ANI matrix 자료 다루기
제목은 ANI matrix(행렬)라고 하였으나 실제로는 dataframe 자료를 다루는 기술에 관하여 작성한 글이다. 실습용 파일 ani_r_exercise.zip을 다운로드하여 압축을 해제한다. RefSeq genome 51개를 pyani(blast)로 처리하여 얻은 결과 중 일부 파일과 newick tree 및 accession 관련 파일이 포함되어 있다.
$ unzip ANI_R_exercise.zip
Archive: ani_r_exercise.zip
inflating: ANIb_percentage_identity.tab
inflating: ANIb_alignment_coverage.tab
inflating: accession-strain.csv
inflating: ezTree_51_pfam.nwk
inflating: accessions.txt
해들리 위캠이 권하는 R 스타일 가이드에 의하면 할당문에는 '=' 말고 '←'를 쓰라고 권장하는데 이는 나의 습관과는 배치된다. 취향의 문제니까 별로 중요하지는 않다. 대부분의 프로그래밍 언어에서 '='를 쓴다는 현실을 잊지 말도록 하자.
Use <-, not =, for assignment.
데이터 파일을 읽어들이고 기본 점검하기
ANI(percentage identity) 계산 결과 파일을 읽어서 ani 데이터프레임을 만든다.
> ani = read.table("ANIb_percentage_identity.tab", sep="\t", header=T, row.names=1)
매트릭스가 아니라 dRep의 Ndb.csv 파일과 같이 <id1>,<id2>,<value> 형태의 파일이라면 임포트한 뒤 reshape 라이브러리를 사용하여 매트릭스로 전환하도록 한다(참조글).
pyani 결과물에서 행과 열의 이름은 이미 같은 순서이다. 굳이 확인하고 싶다면 다음과 같이 한다.
> x = colnames(ani) > y = rownames(ani) > identical(x, y) [1] TRUE
데이터프레임의 대칭 구조가 아니라면 원본과 트랜스포즈한 것을 더한 뒤 2로 나누는 방법을 택하여 평균값을 취하게 만든다. t() 함수를 거친 데이터프레임은 행렬이 되지만, 이를 다시 데이터프레임과 더하면 결과적으로는 데이터프레임이 된다.
> isSymmetric(as.matrix(ani))
[1] FALSE
> ani = (ani + t(ani))/2
> isSymmetric(as.matrix(ani))
[1] TRUE
# if statement를 이용하여 테스트와 대칭 전환을 한 번에 수행
> if (isSymmetric(as.matrix(ani))) {
print("Dataframe ani is symmmetric")
} else {
ani = (ani + t(ani))/2
print("Testing symmetry after conversion...")
isSymmetric(as.matrix(ani))
}
계층적 클러스터링, 시각화 및 트리 자료의 간단한 조작
클러스터링 자체는 R의 기본 기능에 포함된 hclust() 함수로 구현된다. 그러나 트리 자료를 파일로 입출력하거나 정렬된 label을 벡터로 반환하는 등의 작업을 하려면 ape 패키지(홈페이지 활용 설명서)가 필요하다. 물론 뒤에서 설명할 phylogram 패키지(소개의 글)도 있다! ape에서는 표준으로 여겨지는 phylo 객체를 사용하지만 phylogram에서는 dendrogram 객체를 사용한다.
> library(ape)
> fit = hclust(dist(as.matrix(ani)),method="average")
> plot(fit,hang=-1,cex=.7,main="UPGMA Clustering")
# plot(fit)과 plot(tree)는 트리를 그리는 방향이 다르다.
> tree = as.phylo(fit)
> plot(tree,cex=0.7)
> class(tree)
[1] "phylo"
> write.tree(phy=tree,file="ANIb_UPGMA.newick")
> tree # 트리 형태에 대한 설명
Phylogenetic tree with 51 tips and 50 internal nodes.
Tip labels:
GCF_000146875.3, GCF_001956155.1, GCF_000597985.1, GCF_000686825.1, ...
Rooted; includes branch lengths.
> tree$tip.label # Leaf label 표시
# Newick format의 트리 파일을 읽어들이기
> my_tree = ape::read.tree("ezTree_51_pfam.nwk")
> plot(my_tree, cex=0.5)
> is.rooted(my_tree)
[1] FALSE
ezTree_51_pfam.nwk은 unrooted tree이다. ape 패키지의 root() 함수를 사용하면 사용자가 지정한 outgroup이나 노드를 기준으로 rerooting을 할 수 있다. 그러나 간단하게 midpoint rooting을 하려면 phytools 패키지의 midpoint.root()함수를 쓰는 것이 편리하다.
> library(phytools) > my_tree = midpoint.root(my_tree) > is.rooted(my_tree) [1] TRUE > write.tree(phy=my_tree,file="ezTree_51_pfam_rooted.nwk")
클러스터링은 행과 열 중 어느 것을 대상으로 이루어진 것일까? ANI dataframe은 행과 열의 라벨이 동일해서 언뜻 쉽게 파악이 되지 않는다. 정답은 row 단위로 계층적 클러스터링이 되었다는 것이다. R의 데이터프레임에서 개별 자료는 row 단위로 위치한다는 것을 상기하면 쉽게 이해가 될 것이다. 다음을 실행하여 row label을 변경한 뒤 확인해 보자.
> rownames(ani) = gsub("^GCF_", "ROW_", rownames(pid))
> fit = hclust(dist(as.matrix(ani)),method="average")
> plot(fit,hang=-1,cex=.8,main="UPGMA Clustering with row names changed")
클러스터링을 했다고 하여 원본 데이터프레임의 자료 순서가 바뀌지는 않는다.데이터프레임의 행과 열도 클러스터링 결과에 맞추어 재정렬하는 방법을 알아보자.
> label_sorted = fit$labels[c(fit$order)] > ani_sorted = ani[, label_sorted] > ani_sorted = ani_sorted[label_sorted, ] > write.table(ani_sorted, "ANIb_percentage_identity_clustered.csv", sep=",", quote=F)
균주 이름이 들어간 트리를 만들어 보자(데이터프레임 라벨 수정)
현재 각 샘플의 식별자(행과 열의 이름)은 “GCF_000146875.3” 형태의 RefSeq assembly accession이라서 실제로 어떤 균주인지 알기가 어렵다. 원본 자료에서 쓰였던 assembly accession과 균주명을 서로 연결한 텍스트 파일('accession_strain.csv')이 있다면 이를 R에서 읽어들인 후 row label과 바꾼 다음 트리를 만들면 된다. ncbi-genome-download를 '--dry-run' 파라미터와 함께 실행하면 이러한 파일을 쉽게 만들 수 있다.
$ cat accessions.txt
GCF_000146875.3
GCF_001956155.1
GCF_000597985.1
...
$ ncbi-genome-download -A accessions.txt bacteria --dry-run | \
awk -F"\t" '/^GCF/{printf "%s,%s %s\n", $1, $2, $3}' > \
accession_strain.csv
accession_strain.csv 파일은 아쉽지만 수정을 거쳐야 한다. 왜냐하면 두번째 컬럼(organism name) 값의 끝에 strain이 포함된 것이 꽤 있기 때문이다. 이는 유전체 정보를 제출한 사람의 잘못이라고 할 수 있다. 멋진 스크립트를 짜서 이를 수정할 수도 있지만 몇 줄 되지 않는다면 적당히 편집기에서 고치는 것이 나을 것이다. 수정 완료된 accession_strain.csv 파일은 다음과 같아야 한다.
... GCF_000146875.3,Paenibacillus polymyxa E681 GCF_002894905.1,Paenibacillus sp. F4 GCF_000164985.3,Paenibacillus polymyxa SC2 ...
이미 대칭 상태가 되도록 처리를 마친 ani 데이터프레임의 row 라벨을 균주명으로 치환한 ani_s를 생성한 뒤 클러스터링을 해 보자. 아래에 보인 코드의 for{} 블록 내부에 있는 row.names() 함수를 names()로 바꾸면 컬럼 라벨을 바꾸는 효과가 있다. Named vector를 이용하는 매우 유용한 예제이므로 잘 익혀두기 바란다. 바꾸기 전의 라벨이 key 벡터에 들어있고, 바꾸고 나서의 라벨은 names(key)로 지정함을 잊지 말자.
> data = read.table("accession-strain.csv",sep=",",stringsAsFactors=F)
> key = data[,1] # GCF_000463565.1
> names(key) = data[,2] # Paenibacillus polymyxa WLY78
> key
Paenibacillus polymyxa WLY78 Paenibacillus polymyxa DSM 365
"GCF_000463565.1" "GCF_000714835.1"
...
> ani_s = ani
> for (i in row.names(ani_s)) {
temp = names(key)[key==i]
row.names(ani_s)[row.names(ani_s)==i] = temp
}
> fit_s = hclust(dist(as.matrix(ani_s)),method="average")
> plot(fit_s,hang=-1,cex=.7,main="UPGMA Clustering with strain names")
글씨가 많아져서 약간 어수선하지만 균주 정보가 트리에 표시된 것을 볼 수 있다.
균주들을 클러스터 단위로 나누기
각 클러스터에 균주들이 어떻게 포함되는지를 알아보고 그 중에서 하나를 골라 이것으로만 구성되는 ANI 데이터 서브셋을 만들어 보자. 먼저 클러스터 결과를 화면에 띄워 놓아야 한다.
> plot(fit,hang=-1,cex=.8,main="UPGMA Clustering") # 5개의 클러스터로 나누어 보자. > groups = cutree(fit,k=5) # heigt(h)를 기준으로 자를 수도 있다. # 빨간색 상자 5개가 나타난다. 실제로 분할을 해 보자. > table(groups) groups 1 2 3 4 5 21 20 5 2 3 # 1번 그룹에 속하는 멤버는? > names(groups[groups==1]) [1] "GCF_000146875.3" "GCF_001956155.1" "GCF_000686825.1" "GCF_001709075.1" [5] "GCF_900116035.1" "GCF_001707685.1" "GCF_001709135.1" "GCF_000463565.1" [9] "GCF_000520775.1" "GCF_000714835.1" "GCF_001956235.1" "GCF_000507205.3" [13] "GCF_001272655.2" "GCF_001719045.1" "GCF_001593085.1" "GCF_001922145.1" [17] "GCF_002916985.1" "GCF_002894905.1" "GCF_001955935.1" "GCF_001956115.1" [21] "GCF_001956225.1"
1번 그룹 멤버를 벡터로 저장한 다음 ani 데이터프레임에서 이에 해당하는 부분을 뽑아낸 서브셋을 만들어 보자. 데이터가 갖는 조건이 아니라 행 또는 열의 라벨을 이용하여 추출하는 것임에 유의하자.
# 추출할 행/열 라벨을 우선 벡터 x에 지정한다.
> x = names(groups[groups==1])
# 추출할 균주명이 별도의 텍스트 파일('list.txt')에 있다면 readsLines() 함수로 읽어들인다.
# list.txt 파일의 마지막 줄 다음에 줄바꿈이 없으면 불평을 할 것이다.
# readLines()가 밑줄('_')을 자동으로 공백으로 바꾸지 않게 하려면?
> x = readLines("list.txt")
# subset() 함수로 컬럼을 먼저 뽑아내고...
> ani_sub = subset(ani, select=x)
> dim(ani_sub)
[1] 51 21
# 이어서 row를 뽑아낸다.
> ani_sub = subset(ani_sub, rownames(ani_sub) %in% x)
> dim(ani_sub)
[1] 21 21
> write.table(ani_sub, "ANIb_percentage_identity_subset.csv", sep=",", quote=F)
subset() 함수는 컬럼에 대해서 더 친절한 것 같다. 데이터프레임에서 이름에 의해 컬럼을 선택하는 방법은 이것 외에도 몇 가지가 더 있다. 2015년에 썼던 글 [하루에 한 R] 특정 조건에 맞는 row만 출력하기를 돌아보며 잠시 복습을 하자.
Heatmap 그리기
다음의 자료를 통해 영감을 얻었다!
Heatmap in R: Statistics and interactive visualization
한동안 gplots 패키지의 heatmap.2() 함수를 즐겨 사용하였으나 위 문서를 통해 ComplexHeatmap 패키지를 접하고 완전히 이리로 전향하기로 하였다. 기존에 작성한 문서로는 Data visualization이 있다. 여기에서는 ANI 데이터프레임에 대한 heatmap을 만들면서 column은 자체적으로 클러스터링하되 row는 외부에서 임포트한 자료를 사용하는 방법을 설명한다. 아울러서 heatmap에 표시된 label을 자유롭게 바꾸는 방법도 알아보도록 한다.
phylogram으로 외부 트리 읽어들이기
ape 패키지의 read.tree() 함수로 읽어들인 phylo 객체를 as.dendrogram() 함수로 전환해도 된다.
> library(phylogram) > tree.2 = read.dendrogram(file="ezTree_51_pfam_rooted.nwk") # dendrogram class에서 tip label 추출하기 > labels(tree.2) > plot(tree.2)
Heatmap 그리기
우선 ani 데이터프레임을 자체적으로 클러스터링하여 heatmap을 만들어 보자. gplots heatmap.2()는 레이아웃을 수정하지 않으면 보기에 좀 불편할 것이다. 자세한 사항은 이전에 쓴 글 Data visualization을 참고하라. 반면 ComplexHeatmap Heatmap()은 특별히 레이아웃을 건드리지 않아도 라벨이 길다고 하여 경계 바깥으로 잘리거나 하지 않아서 매우 좋다.
> library(gplots)
> heatmap.2(as.matrix(ani),col=greenred(10),trace="none",scale="none",dendrogram=c("row"))
> library(ComplexHeatmap)
> Heatmap(as.matrix(ani), name="ANI", row_names_gp = gpar(fontsize = 5), column_names_gp = gpar(fontsize = 5))
 이번에는 row와 column을 서로 다른 방식으로 클러스터링해 보자. row는 임포트한 트리(ezTree_51_pfam_rooted.nwk, dendrogram 객체여야 함)를 기준으로 정렬하되 column은 ani 데이터프레임의 값으로 자체적인 클러스터링을 적용한다. 실제로 그려보니 별로 예쁘질 않다.
이번에는 row와 column을 서로 다른 방식으로 클러스터링해 보자. row는 임포트한 트리(ezTree_51_pfam_rooted.nwk, dendrogram 객체여야 함)를 기준으로 정렬하되 column은 ani 데이터프레임의 값으로 자체적인 클러스터링을 적용한다. 실제로 그려보니 별로 예쁘질 않다.
> Heatmap(as.matrix(ani), name="ANI", cluster_rows = tree.2, row_names_gp = gpar(fontsize = 5), column_names_gp = gpar(fontsize = 5))
row와 column 전부 tree.2를 기준으로 정렬해 보자.
> Heatmap(as.matrix(ani), name="ANI", cluster_rows = tree.2, cluster_columns = tree.2, row_names_gp = gpar(fontsize = 5), column_names_gp = gpar(fontsize = 5))
Label을 자유롭게 표시하기
위에서 보인 ani 데이터프레임의 라벨 치환 사례에서는 accession-strain.csv 파일로부터 data 데이터프레임을 만들고, 이로부터 key라는 named vector를 생성하여 사용했었다. 그러나 Heatmap() 함수에서는 각 축의 고유한 라벨은 그대로 두되 그림으로 나타낼 라벨만 원하는 형태로 바꿀 수 있다. 단, data 데이터프레임으로부터 아까의 것과는 반대 구조를 갖는 named vector(key.2)를 만들어야 한다. 편의상 row label만 실제 균주의 full name으로 바꾸어서 heatmap을 그려 보자.
> key.2 = data[,2] # Paenibacillus polymyxa WLY78
> names(key.2) = data[,1] # GCF_000463565.1
> key.2
GCF_000463565.1 GCF_000714835.1
"Paenibacillus polymyxa WLY78" "Paenibacillus polymyxa DSM 365"
...
> Heatmap(as.matrix(ani), name = "ANI", row_labels = key.2[rownames(ani)], row_names_gp = gpar(fontsize = 5), column_names_gp = gpar(fontsize = 5))
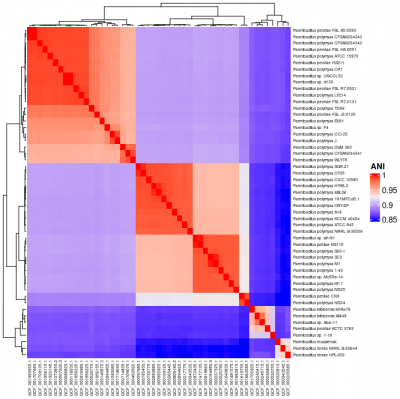 ComplexHeatmap의 활용법을 자세히 알아보려면 Complete Reference를 참고하기 바란다.
ComplexHeatmap의 활용법을 자세히 알아보려면 Complete Reference를 참고하기 바란다.